Miser sur le potentiel de l’IA et des omiques
Les progrès technologiques en génomique permettent de faire évoluer constamment et rapidement notre compréhension des associations génotype- phénotype. Un levier reconnu comme garantissant la découverte de nouveaux diagnostics consiste dès lors en la réé-valuation des signes cliniques des malades et passe par la constitution d’une liste de phénotypes (signes morphologiques, physiologiques…) aussi exhaustive que possible, intégrant les nouveaux symptômes lorsque ceux-ci se présentent. Le recueil des descriptions phénotypiques des patients sous une forme normée et exploitable informatiquement (ontologies) est ainsi nécessaire et néanmoins particulièrement consommateur de temps, ce qui en limite l’usage en vie réelle. C’est dans un tel contexte qu’apparaît tout le potentiel des transformeurs et grands modèles de langage (LLM), capables de révolutionner le traitement automatique du langage naturel en offrant des performances sans précédent en termes de compréhension, de génération et d’ana-lyse du langage. En effet, ces modèles s’avèrent particulièrement puissants pour extraire et prédire des listes de phénotypes pertinents à partir des données textuelles de comptes-rendus cliniques2. Leur adaptation et implémentation dans le contexte particulier des maladies rares en langue française permettent aujourd’hui d’envisager l’établissement systématique d’un tableau clinique complet pour l’ensemble des patients sans surcharge de travail pour les généticiens cliniciens. Avec une structuration adéquate des données, ils permettraient également une actualisation automatique de ce tableau clinique tout au long du parcours de soins des individus, sui-vant ainsi l’histoire naturelle de la maladie et aidant ce faisant le diagnostic différentiel et, par extension, la détermination de la cause moléculaire.
En complément de l’évaluation clinique des patients, un levier pour résoudre davantage d’impasses dia-gnostiques réside dans l’analyse complémentaire de multiples niveaux moléculaires dans une approche dite « multiomique ». Cette approche biosystémique permet de corréler variants génétiques et effets transcriptionnels, protéiques ou métaboliques. Le séquençage de transcrits permet par exemple d’identifier les conséquences des variants géné-tiques sur la suite des processus cellulaires, telles une sur- ou sous-expression anormale d’un gène ou un épissage aberrant, impactant le nombre ou la structure des protéines produites.
Le croisement des résultats de l’extraction automati-sée de phénotypes et d’éventuelles données omiques complémentaires avec une ré-analyse systématique à l’aune de connaissances mondiales actualisées des données de séquençage ADN préexistantes permettrait ainsi une valorisation maximale des données produites au sein des établissements. De telles ré-analyses systématisées nécessitent cependant une structuration efficace des données de santé et le développement d’outils innovants dédiés pour optimiser le rendement diagnostique tout en limitant la surcharge de travail pour les biologistes et cliniciens.
Des projets structurants dans le Grand Ouest
Deux projets pilotes ont été initiés au sein du groupement de coopération sanitaire des Hôpitaux universitaires Grand Ouest (GCS HUGO). Leur objectif commun est de relever ces défis en s’appuyant sur les technologies omiques, l’intelligence artificielle et les données massives. Ces initiatives visent à mieux accompagner les personnes atteintes de maladies rares d’origine génétique. Le premier projet, Diagho, est un portail d’interprétation génomique ; le second, HUGO-RD, se concentre sur l’extraction et la structuration automatisées des informations cliniques des comptes-rendus médicaux.
Diagho, le portail d’interprétation génomique du Grand Ouest
Le portail d’interprétation génomique du Grand Ouest, Diagho, est né d’une dynamique régionale forte visant à créer un outil académique commun d’aide à l’interprétation de variants génétiques dans un cadre diagnostique afin de mutualiser les coûts, d’homogénéiser les pratiques et d’aider le partage d’expertises au sein de l’interrégion. Son but est de proposer une interface utilisateur permettant de filtrer efficacement et de mettre en corrélation les données des différentes omiques afin d’optimiser le rendement diagnostique et le temps alloué aux examens. En simplifiant le déroulement des interprétations, les biologistes peuvent traiter plus de dossiers, accorder davantage de temps aux cas complexes et réaliser ainsi de plus nombreux diagnostics, limitant par conséquent l’errance d’un maximum de familles. Avec un tel système d’aide à l’interprétation, il devient également possible d’envisager la systématisation du dépistage néonatal par analyse génomique en première intention comme proposé par le projet Perigenomed.
DIAGHO POUR UN DÉPISTAGE NÉONATAL PLUS PERFORMANT
Porté par la FHU Translad, le projet Perigenomed ambitionne de révolutionner le dépistage néonatal (DNN) en France grâce au séquençage génomique à haut débit. Enciblant les maladies rares traitables ou actionnables à un stade précoce, il vise à améliorer le pronostic et le confort de vie des nouveau-nés et à développer une filière innovante pour des solutions diagnostiques et thérapeutiques. L’objectif est de fournir aux parents des résultats en moins de quatre semaines. Dans le Grand Ouest, les CHU de Rennes, Nantes et Angers, partenaires du projet, mèneront l’interprétation moléculaire sur le portail Diagho, depuis l’analyse des variations jusqu’à la génération des rapports.
Outre cette simplification des pratiques, le portail Diagho intègre une fonctionnalité de ré-analyse des diagnostics négatifs, au travers de méthodes de surveillance automatisées des connaissances scientifiques tout en prenant compte de l’évolution des tableaux cliniques des patients, améliorant ainsi le rendement diagnostique, avec un investissement en ressource médicale réduit.
En permettant l’analyse de cohortes sur différents types de données ainsi que le partage de procédures, de stratégies de filtrage et de conclusions d’interprétations de variants, Diagho se positionne au-delà du soin, comme outil d’aide à la recherche et favorise la découverte de nouvelles associations gènes-maladies et leurs partages.
Enfin, ce projet vise également l’enrichissement de bases de connaissances nationales via un verse-ment de données simplifié vers le collecteur analyseur de données (CAD) du plan France Médecine génomique 2025, ou directement vers des bases expertes comme la base d’oncogénétique FrOG ou la base nationale de variations du nombre de copies BANCCO+.
HUGO-RD, le chaînon manquant entre génétique clinique et moléculaire
Le second programme, HUGO-RD, projet pilote du Ouest Data Hub, a quant à lui pour objectif l’automatisation de la collecte des données phénotypiques des patients depuis les comptes-rendus médicaux. Ce projet, développé en partenariat avec un institut de recherche, vise à développer des modèles de langage adaptés au domaine de la génétique médicale en français, en exploitant les avancées récentes en intelligence artificielle.
De façon concrète, ces modèles tirent parti de leur capacité à apprendre « sur le tas » (in-context learning), c’est-à-dire à résoudre des tâches complexes en se basant sur des exemples fournis. Pour atteindre ces objectifs, il est essentiel de constituer des corpus de données adaptés pour garantir que les modèles entraînés soient à la fois pertinents, précis et capables de s’adapter aux défis spécifiques du domaine de la génétique médicale. Ainsi, nous avons constitué un corpus unique de plus de 60 000 comptes-rendus médicaux provenant des CHU de Rennes, Nantes et Angers3. Cet ensemble de données soigneusement sélectionnées et structurées couvre la terminologie spécialisée, les concepts cliniques et les scénarios variés rencontrés en génétique médicale. Il constitue la base indispensable pour entraîner des modèles capables de fournir des analyses précises, de répondre aux besoins spécifiques des cliniciens et d’améliorer la prise en charge des patients atteints de maladies rares.
Parmi les modèles de pointe utilisés, nous exploitons particulièrement PhenoBERT, un modèle spécialisé dans l’identification des phénotypes. Par ailleurs, des systèmes combinant génération de texte et récupération d’informations, appelés RAG (Retrieval-Augmented Generation)4, ont été testés avec succès. Ces systèmes fonctionnent comme des assistants intelligents qui consultent des bases de données spécialisées, telles que des graphes de connaissances ou des ontologies médicales, pour enrichir leurs réponses. Ils permettent ainsi de traiter efficacement des volumes importants d’informations tout en restant précis et contextualisés. En complément, les modèles sont ajustés de manière ciblée (fine-tuning) pour répondre aux besoins spécifiques du domaine et appuyés par le développement d’un outil d’an-notation performant pour créer des ensembles de données pertinents et intégrables dans des workflows d’apprentissage automatique. Cet outil permet à la communauté de bénéficier d’une anno-tation automatique experte des comptes-rendus et d’en extraire les informations essentielles à la compréhension de l’état d’un patient. Déployé sur l’ensemble des sites d’HUGO, il permet de systéma-tiser l’extraction pertinente des informations sous la forme d’un vocabulaire standardisé et structuré, interopérable et utilisable pour des traitements informatiques ultérieurs, et particulièrement en accompagnement d’une réanalyse systématique du génome des patients.
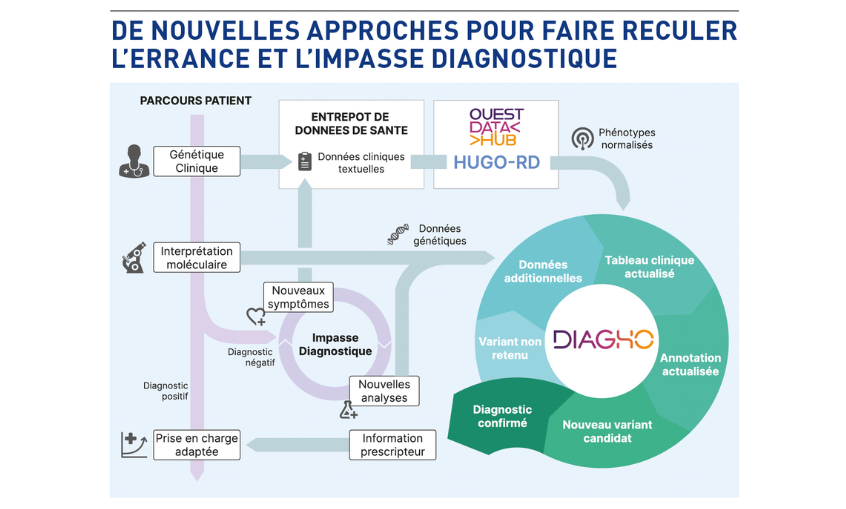
Diagho et HUGO-RD ouvrent la voie à de nouvelles approches pour faire reculer l’errance et l’impasse diagnostiques. Grâce à un chaînage et une structuration des données de santé dans tout le Grand Ouest, il émerge une organisation fédérée et efficiente pour l’ensemble des informations diagnos-tiques dans l’interrégion, incluant les données de séquençage, les données de comptes-rendus médi-caux et les workflows d’interprétation. Il devient dès lors possible de reconstruire les parcours médicaux des patients, de mieux comprendre l’histoire natu-relle des pathologies et de réaliser une ré-analyse systématique des données d’exomes et de génomes négatifs avec des workflows innovants, optimisés et standardisés pour lever un maximum d’impasses diagnostiques tout en gardant la charge minime pour les praticiens.
